案例一 水中溶菌酶
这个示例将指导新用户完成设置模拟系统的过程,该系统在一盒水中含有蛋白质 (溶菌酶) 和离子。每个步骤都将包含输入和输出的说明,使用一般使用的典型设置。
整个教程的步骤如下: 
准备拓扑文件
PDB检索
在模拟开始前下载大分子pdb文件,通常是在RCSB数据库官网下载,在本教程中,我们将使用鸡蛋清溶菌酶(PDB 代码1AKI)
下载结构后,使用 VMD、Chimera、PyMOL 等查看程序可视化结构。
去水
接下来要处理大分子文件,删除水分子(PDB 文件中的 HOH),请使用 vi、emacs (Linux/Mac) 或记事本 (Windows) 等纯文本编辑器,不要使用文字处理软件!
这里推荐统一使用VScode,配置工作路径和gmx插件后使用。
或者使用以下gmx命令:
grep -v HOH 1aki.pdb > 1AKI_clean.pdb使用Pymol等软件去水同样可以!
并不是每次都要去水(例如有紧密结合或其他功能性活性位点水分子的情况),本案例中不需要水分子。
生成拓扑
现在,水分子已经去除,我们已经验证了所有必要的原子都存在,PDB文件应该只包含蛋白质原子,并准备好输入到第一个GROMACS模块pdb2gmx中。pdb2gmx 的目的是生成三个文件:
- 分子的拓扑结构
- 位置约束文件
- 后处理的结构文件
拓扑(默认为 topol.top)包含在仿真中定义分子所需的所有信息。此信息包括非键合参数(原子类型和电荷)以及键合参数(键、角度和二面体)。生成拓扑后,我们将更详细地查看拓扑。
通过发出以下命令来执行 pdb2gmx:
gmx pdb2gmx -f 1AKI_clean.pdb -o 1AKI_processed.gro -water spce该结构将由 pdb2gmx 处理,并且系统将提示您选择一个力场:
Select the Force Field:
From '/usr/local/gromacs/share/gromacs/top':
1: AMBER03 protein, nucleic AMBER94 (Duan et al., J. Comp. Chem. 24, 1999-2012, 2003)
2: AMBER94 force field (Cornell et al., JACS 117, 5179-5197, 1995)
3: AMBER96 protein, nucleic AMBER94 (Kollman et al., Acc. Chem. Res. 29, 461-469, 1996)
4: AMBER99 protein, nucleic AMBER94 (Wang et al., J. Comp. Chem. 21, 1049-1074, 2000)
5: AMBER99SB protein, nucleic AMBER94 (Hornak et al., Proteins 65, 712-725, 2006)
6: AMBER99SB-ILDN protein, nucleic AMBER94 (Lindorff-Larsen et al., Proteins 78, 1950-58, 2010)
7: AMBERGS force field (Garcia & Sanbonmatsu, PNAS 99, 2782-2787, 2002)
8: CHARMM27 all-atom force field (CHARM22 plus CMAP for proteins)
9: GROMOS96 43a1 force field
10: GROMOS96 43a2 force field (improved alkane dihedrals)
11: GROMOS96 45a3 force field (Schuler JCC 2001 22 1205)
12: GROMOS96 53a5 force field (JCC 2004 vol 25 pag 1656)
13: GROMOS96 53a6 force field (JCC 2004 vol 25 pag 1656)
14: GROMOS96 54a7 force field (Eur. Biophys. J. (2011), 40,, 843-856, DOI: 10.1007/s00249-011-0700-9)
15: OPLS-AA/L all-atom force field (2001 aminoacid dihedrals)force 字段将包含将写入拓扑的信息。您应该始终仔细阅读每个力场并确定哪个最适合您的情况。
在本教程中,我们将使用全原子OPLS力场,因此在命令提示符下键入15,然后按Enter。
可选参数
还有许多其他选项可以传递给pdb2gmx这里列出了一些常用的:
-ignh:忽略 PDB 文件中的 H 原子;特别适用于核磁共振结构。否则,如果存在 H 原子,则它们必须按照 GROMACS 中的力场期望的命名方式命名。存在不同的约定,因此处理 H 原子有时会令人头疼!如果您需要保留初始 H 坐标,但需要重命名,那么 Linux sed 命令是您的好帮手。
-ter:交互式地为 N 端和 C 端分配电荷态。
-inter:交互式地为 Glu、Asp、Lys、Arg 和 His 分配电荷态;选择哪些 Cys 参与二硫键。
结果分析
您现在已经生成了三个新文件:1AKI_processed.gro、topol.top和 posre.itp。1AKI_processed.gro 是一个 GROMACS 格式的结构文件,其中包含力场中定义的所有原子(即,H 原子已添加到蛋白质中的氨基酸中)。topol.top 文件是系统拓扑(稍后会详细介绍)。posre.itp 文件包含用于约束重原子位置的信息(稍后将详细介绍)。
最后一点:许多用户认为 .gro 文件是必需的,其实并不是。GROMACS 可以处理许多不同的文件格式,其中 .gro 只是写入坐标文件的命令的默认值。这是一种非常紧凑的格式,但精度有限。例如,如果您更喜欢使用 PDB 格式,您需要做的就是指定一个带有 .pdb 扩展名的适当文件名作为输出。pdb2gmx 的目的是生成符合力场的拓扑;output 结构在很大程度上是此目的的副作用,旨在方便用户。格式可以是您喜欢的任何内容。
检查拓扑
topol.top
输出拓扑 topol.top 文件,可以使用纯文本编辑器打开。;符号表示为注释行。
#include "oplsaa.ff/forcefield.itp"此行调用 OPLS-AA 力场中的参数,它位于文件的开头,表示所有后续参数都来自此力场。
下一个重要行是[ moleculetype ]:
[ moleculetype ]
; Name nrexcl
Protein_A 3名称 Protein_A 定义了分子名称,因为蛋白质在 PDB 文件中被标记为链 A 的事实。绑定邻域有 3 个排除项。
下一节[ atoms ]定义了蛋白中的氨基酸序列,信息以列的形式显示:
[ atoms ]
; nr type resnr residue atom cgnr charge mass typeB chargeB massB
; residue 1 LYS rtp LYSH q +2.0
1 opls_287 1 LYS N 1 -0.3 14.0067 ; qtot -0.3
2 opls_290 1 LYS H1 1 0.33 1.008 ; qtot 0.03
3 opls_290 1 LYS H2 1 0.33 1.008 ; qtot 0.36
4 opls_290 1 LYS H3 1 0.33 1.008 ; qtot 0.69
5 opls_293B 1 LYS CA 1 0.25 12.011 ; qtot 0.94
6 opls_140 1 LYS HA 1 0.06 1.008 ; qtot 1本节信息解读如下:
- nr:原子数
- type: 原子型
- resnr:氨基酸残基数
- residue:氨基酸残基名称;请注意,此残基在 PDB 文件中为 “LYS”;使用
.rtp,“LYSH”表示残基是质子化的(中性 pH 值下的主要状态)。 - atom: 原子名称
- cgnr:电荷组编号;电荷组定义整数电荷的单位,它们有助于加快计算速度
- charge:
qtot描述符是分子上电荷的运行总和 - mass:质量
- typeB, chargeB, massB: 用于自由能扰动(此处不讨论)
[ bonds ]是化学键, [ pairs ]原子非键合作用力, [ angles ]表示二键角, [ dihedrals ]表示二面角。
Protein_A 分子型定义到此结束,拓扑文件的其余部分专门用于定义其他分子并提供系统级描述,主要包括位置约束等。
; Include Position restraint file
#ifdef POSRES
#include "posre.itp"
#endif下一个分子型(默认)是溶剂,在本例中为 SPC/E 水。其他典型的水选择包括 SPC、TIP3P 和 TIP4P。我们通过将 -water spce 传递给 pdb2gmx 来选择它。
水也可以使用 1000 kJ mol-1 nm-2 的力常数 (kpr) 进行位置约束。
; Include water topology
#include "oplsaa.ff/spce.itp"
#ifdef POSRES_WATER
; Position restraint for each water oxygen
[ position_restraints ]
; i funct fcx fcy fcz
1 1 1000 1000 1000
#endif接下来包括离子参数:
; Include generic topology for ions
#include "oplsaa.ff/ions.itp"最后是系统级定义,该指令给出了在仿真期间将写入输出文件的系统的名称,该指令列出了系统中的所有分子:
[ system ]
; Name
LYSOZYME
[ molecules ]
; Compound #mols
Protein_A 1关于该指令的一些关键说明:
- 列出的分子的顺序必须与坐标(在本例中为
.gro)文件中的分子顺序完全匹配。 - 列出的名称必须与每个物种的名称相匹配,而不是残留物名称或其他任何内容。
- 如果这些具体要求没有符合条件, grompp 将会抛出致命错误,例如名称不匹配、未找到分子或许多其他错误。
posre.itp
posre.itp 是位置限制文件,由 pdb2gmx 生成,它定义了一个力常数,用于在平衡过程中将原子保持在原位。
示例:
[ position_restraints ]
; atom type fx fy fz
1 1 1000 1000 1000
5 1 1000 1000 1000
7 1 1000 1000 1000
10 1 1000 1000 1000
13 1 1000 1000 1000定义盒子并溶剂化
这个例子中,我们将模拟一个简单的水性系统,可以在不同的溶剂中模拟蛋白质和其他分子,前提是所有相关物种都有良好的参数。
定义盒子
定义盒子并在其中填充溶剂有两个步骤:
- 使用
editconf模块定义盒子尺寸 - 使用
solvate模块(以前称为 genbox)将盒子装满水。
在本教程中,我们将使用一个简单的立方框作为基本单元,随着您对周期性边界条件和框类型越来越熟悉,可以使用菱形十二面体,因为它的体积是相同周期距离的立方框的 ~71%,从而节省了需要添加以溶解蛋白质的水分子数量。
让我们用editconf定义这个盒子:
gmx editconf -f 1AKI_processed.gro -o 1AKI_newbox.gro -c -d 1.0 -bt cubic上述命令将蛋白质在框 (-c) 中居中,并将其放置在距框边缘至少 1.0 nm (-d 1.0) 的位置。框类型定义为多维数据集 (-bt cubic)。到框边缘的距离是一个重要参数。由于我们将使用周期性边界条件,因此必须满足最小图像约定。也就是说,蛋白质永远不应该看到它的周期性图像,否则计算的力将是虚假的。指定 1.0 nm 的溶质盒距离意味着蛋白质的任意两个周期性图像之间至少有 2.0 nm。这个距离对于模拟中常用的几乎任何截止方案都足够了。
溶剂化
现在我们已经定义了一个盒子,我们可以用溶剂(水)填充它。溶剂化是使用溶剂化物完成的:
gmx solvate -cp 1AKI_newbox.gro -cs spc216.gro -o 1AKI_solv.gro -p topol.top蛋白质的配置 (-cp) 包含在上一个editconf步骤的输出中,溶剂的配置 (-cs) 是标准 GROMACS 安装的一部分。我们使用的是spc216.gro,这是一个通用的平衡 3 点溶剂模型。您可以使用spc216.gro作为 SPC、SPC/E 或 TIP3P 水的溶剂配置,因为它们都是三点水模型。输出名为1AKI_solv.gro,我们告诉solvate拓扑文件的名称 (topol.top) 以便对其进行修改。
请注意对 topol.top 指令的更改:
[ molecules ]
; Compound #mols
Protein_A 1
SOL 10832solvate所做的是跟踪它添加了多少水分子,然后将其写入您的拓扑以反映所做的更改。请注意,如果您使用任何其他(非水)溶剂,solvate不会对您的拓扑进行这些更改!它与更新水分子的兼容性是硬编码的。
添加离子
我们现在有一个包含带电蛋白质的溶剂化系统。pdb2gmx 的输出告诉我们,该蛋白质的净电荷为 +8e(基于其氨基酸组成)。如果您在 pdb2gmx 输出中错过了此信息,查看 topol.top 中指令的最后一行;它应该读作(部分)“QTOT 8”。由于生命不以净电荷存在,因此我们必须将离子添加到我们的系统中。
在 GROMACS 中添加离子的工具称为genion。它可以读取拓扑结构,并用用户指定的离子替换水分子。该输入称为运行输入文件,其扩展名为 .tpr;此文件由 GROMACS grompp 模块 (GROMACS pre-processor) 生成,稍后在运行第一次模拟时也将使用该文件。 grompp 所做的是处理坐标文件和拓扑(描述分子)以生成原子级输入 .tpr。.tpr 文件包含系统中所有原子的所有参数。
构建mdp文件
构建下列ions.mdp文件:
; ions.mdp - used as input into grompp to generate ions.tpr
; Parameters describing what to do, when to stop and what to save
integrator = steep ; Algorithm (steep = steepest descent minimization)
emtol = 1000.0 ; Stop minimization when the maximum force < 1000.0 kJ/mol/nm
emstep = 0.01 ; Minimization step size
nsteps = 50000 ; Maximum number of (minimization) steps to perform
; Parameters describing how to find the neighbors of each atom and how to calculate the interactions
nstlist = 1 ; Frequency to update the neighbor list and long range forces
cutoff-scheme = Verlet ; Buffered neighbor searching
ns_type = grid ; Method to determine neighbor list (simple, grid)
coulombtype = cutoff ; Treatment of long range electrostatic interactions
rcoulomb = 1.0 ; Short-range electrostatic cut-off
rvdw = 1.0 ; Short-range Van der Waals cut-off
pbc = xyz ; Periodic Boundary Conditions in all 3 dimensions构建tpr文件
要生成带有 grompp 的 .tpr 文件,我们需要一个扩展名为 .mdp 的额外输入文件;Grompp 会将 .mdp 文件中指定的参数与坐标和拓扑信息进行组合,以生成 .tpr 文件。
.mdp 文件通常用于运行能量最小化或 MD 模拟,但在这种情况下,它仅用于生成系统的原子描述。可以在此处下载示例 .mdp 文件(我们将使用的那个)。
实际上,此步骤中使用的 .mdp 文件可以包含任何合法的参数组合。我通常使用能量最小化脚本,因为它们非常基本,不涉及任何复杂的参数组合。请注意,本教程提供的文件仅用于 OPLS-AA 力场。其他力场的设置(尤其是非接合交互设置)将有所不同。
将 .tpr 文件与以下内容组合在一起:
gmx grompp -f ions.mdp -c 1AKI_solv.gro -p topol.top -o ions.tpr现在我们在二进制文件 ions.tpr 中对系统进行了原子级描述。我们将此文件传递给 genion:
gmx genion -s ions.tpr -o 1AKI_solv_ions.gro -p topol.top -pname NA -nname CL -neutral出现提示时,选择第 13 组 “SOL” 进行嵌入离子,不用离子替换蛋白质的某些部分。
在 genion 命令中,我们提供结构/状态文件 (-s) 作为输入,生成一个 .gro 文件作为输出 (-o),处理拓扑 (-p) 以反映水分子的去除和离子的添加,定义正离子和负离子名称(分别为 -pname 和 -nname),并告诉 genion 通过添加正确数量的负离子(-neutral、 在这种情况下,将添加 8 个 Cl- 离子以抵消蛋白质上的 +8 电荷)。除了通过同时指定 -neutral 和 -conc 选项来中和系统之外,您还可以使用 genion 添加指定浓度的离子。
在 GROMACS 的早期版本中,使用 -pname 和 -nname 指定的离子名称是力场特定的,但在 4.5 版中是标准化的。指定的离子名称始终是所有大写字母的元素符号,这是随后写入拓扑的名称。残基或原子名称可能会也可能不会附加电荷的符号 (+/-),具体取决于力场。请勿在 genion 命令中使用 atom 或 residue 名称,否则在后续步骤中会遇到错误。
此时的topol.top文件[ molecules ]应如下所示:
[ molecules ]
; Compound #mols
Protein_A 1
SOL 10636
CL 8此时的1AKI_solv_ions.gro文件如下: 
能量最小化
溶剂化的电中性系统现已组装完成,在开始动力学之前,我们必须确保系统没有空间冲突或不适当的几何图形。该结构通过称为能量最小化 (EM) 的过程而松弛。
EM 的过程很像离子的添加。我们将再次使用 grompp 将结构、拓扑和仿真参数组装成二进制输入文件 (.tpr),但这一次,我们将通过 GROMACS MD 引擎 mdrun 运行能量最小化,而不是将 .tpr 传递给 genion。
构建mdp文件
新建以下内容的minim.mdp文件:
; minim.mdp - used as input into grompp to generate em.tpr
; Parameters describing what to do, when to stop and what to save
integrator = steep ; Algorithm (steep = steepest descent minimization)
emtol = 1000.0 ; Stop minimization when the maximum force < 1000.0 kJ/mol/nm
emstep = 0.01 ; Minimization step size
nsteps = 50000 ; Maximum number of (minimization) steps to perform
; Parameters describing how to find the neighbors of each atom and how to calculate the interactions
nstlist = 1 ; Frequency to update the neighbor list and long range forces
cutoff-scheme = Verlet ; Buffered neighbor searching
ns_type = grid ; Method to determine neighbor list (simple, grid)
coulombtype = PME ; Treatment of long range electrostatic interactions
rcoulomb = 1.0 ; Short-range electrostatic cut-off
rvdw = 1.0 ; Short-range Van der Waals cut-off
pbc = xyz ; Periodic Boundary Conditions in all 3 dimensions使用以下输入参数文件通过 grompp 组装二进制输入:
gmx grompp -f minim.mdp -c 1AKI_solv_ions.gro -p topol.top -o em.tpr确保在运行 genbox 和 genion 时一直在更新 topol.top 文件,否则您将收到许多令人讨厌的错误消息(“坐标文件中的坐标数量与拓扑不匹配”等)。
执行能量最小化
现在,我们已准备好调用 mdrun 来执行 EM:
gmx mdrun -v -deffnm em-v 字符可以输出 mdrun 的每一步都进度,-deffnm 为将定义输入和输出的文件名。因此,如果您没有将 grompp 输出命名为 “em.tpr”,则必须使用 mdrun -s 标志显式指定其名称。在我们的例子中,我们将得到以下文件:
- em.log:EM 进程的 ASCII 文本日志文件
- em.edr:二进制能量文件
- em.trr:二进制全精度轨迹
- em.gro:能量最小化结构
检验结果
要确定 EM 是否成功,需要评估两个非常重要的因素。第一个是势能(在 EM 过程结束时打印,即使没有 -v)。Epot 应为负数,并且(对于水中的简单蛋白质)约为 10 5-106,具体取决于系统大小和水分子的数量。第二个重要特征是最大力 Fmax,其目标在 minim.mdp 中设置 - “emtol = 1000.0” - 表示目标 Fmax 不大于 1000 kJ mol-1 nm-1。有可能得出一个合理的 E锅,其中 Fmax > emtol。如果发生这种情况,您的系统可能不够稳定,无法进行仿真。评估它可能发生的原因,并可能更改您的最小化参数(积分器、emstep 等)。
我们来做一些分析。em.edr 文件包含 GROMACS 在 EM 期间收集的所有能量项。您可以使用 GROMACS 能量模块分析任何 .edr 文件:
gmx energy -f em.edr -o potential.xvg在提示符下,键入 “10 0” 以选择 Potential (10);0 终止输入。您将看到 Epot 的平均值,并且将写入一个名为 potential.xvg 的文件。要绘制此数据,需要 Xmgrace 绘图工具。生成的图应该看起来像这样,展示了 Epot 的良好、稳定的收敛:
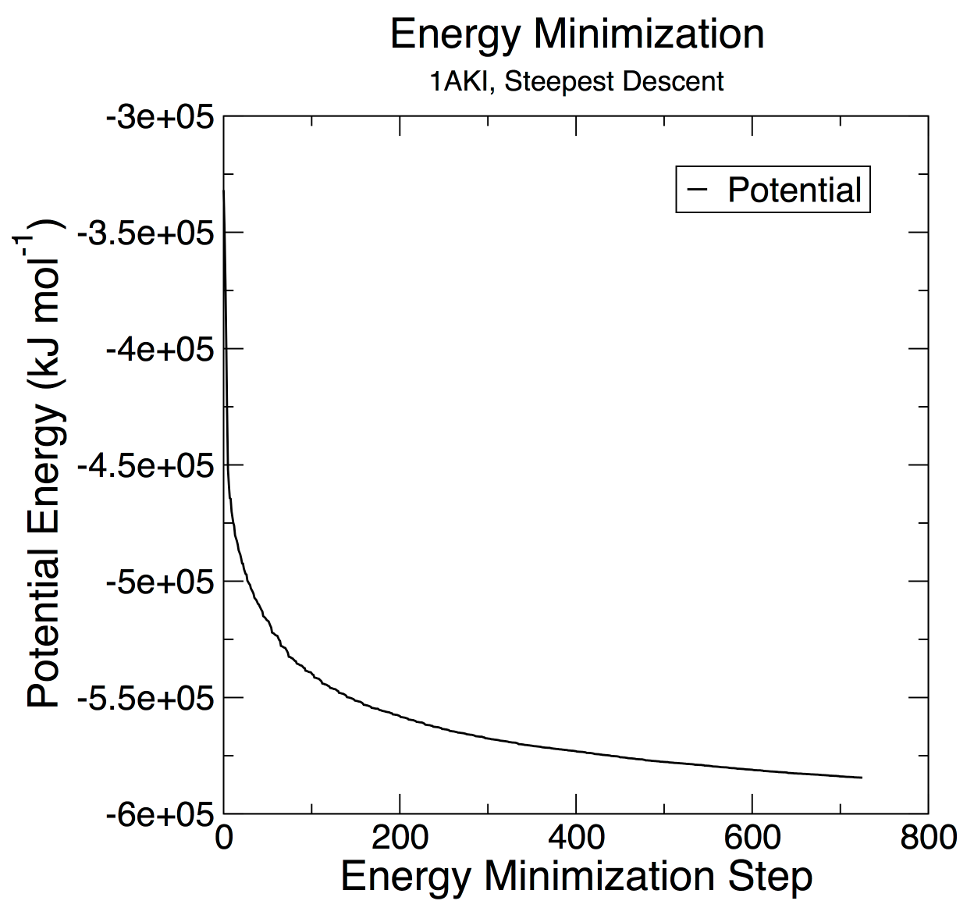
现在我们的系统处于能量最低水平,随后开始动力学模拟部分。
平衡
EM 确保我们在几何形状和溶剂取向方面具有合理的起始结构。要开始真正的动力学,我们必须平衡蛋白质周围的溶剂和离子。如果我们在这一点上尝试无拘无束的动态,系统可能会崩溃。原因是溶剂大部分内部都是优化的,而不一定是溶质的优化。需要将其加热到我们希望模拟的温度,并建立有关溶质(蛋白质)的正确方向。在我们达到正确的温度(基于动能)后,我们将对系统施加压力,直到它达到适当的密度。
还记得很久以前 pdb2gmx 生成的那个 posre.itp 文件吗?我们现在就使用它。posre.itp 的目的是对蛋白质的重原子(任何不是氢的东西)施加位置应变力。允许移动,但前提是克服了大量的能量损失。位置约束的实用性在于,它们允许我们平衡蛋白质周围的溶剂,而不会增加蛋白质结构变化的变量。位置约束的原点 (约束电位为零的坐标) 通过传递给 grompp 的 -r 选项的坐标文件提供。
平衡通常分两个阶段进行。第一阶段在 NVT 系综(恒定的粒子数、体积和温度)下进行。这个系综也被称为 “isothermal -isochoric” 或 “canonical”。这种程序的时间框架取决于系统的内容,但在 NVT 中,系统的温度应该达到所需值的平台。如果温度尚未稳定,则需要额外的时间。通常,50-100 ps 就足够了,我们将为这个练习进行 100 ps 的 NVT 平衡。根据您的机器,这可能需要一段时间(如果在 16 个内核左右并行运行,则不到一个小时)。
温度平衡 NVT
构建mdp文件
构建下列nvt.mdp文件:
title = OPLS Lysozyme NVT equilibration
define = -DPOSRES ; position restrain the protein
; Run parameters
integrator = md ; leap-frog integrator
nsteps = 50000 ; 2 * 50000 = 100 ps
dt = 0.002 ; 2 fs
; Output control
nstxout = 500 ; save coordinates every 1.0 ps
nstvout = 500 ; save velocities every 1.0 ps
nstenergy = 500 ; save energies every 1.0 ps
nstlog = 500 ; update log file every 1.0 ps
; Bond parameters
continuation = no ; first dynamics run
constraint_algorithm = lincs ; holonomic constraints
constraints = h-bonds ; bonds involving H are constrained
lincs_iter = 1 ; accuracy of LINCS
lincs_order = 4 ; also related to accuracy
; Nonbonded settings
cutoff-scheme = Verlet ; Buffered neighbor searching
ns_type = grid ; search neighboring grid cells
nstlist = 10 ; 20 fs, largely irrelevant with Verlet
rcoulomb = 1.0 ; short-range electrostatic cutoff (in nm)
rvdw = 1.0 ; short-range van der Waals cutoff (in nm)
DispCorr = EnerPres ; account for cut-off vdW scheme
; Electrostatics
coulombtype = PME ; Particle Mesh Ewald for long-range electrostatics
pme_order = 4 ; cubic interpolation
fourierspacing = 0.16 ; grid spacing for FFT
; Temperature coupling is on
tcoupl = V-rescale ; modified Berendsen thermostat
tc-grps = Protein Non-Protein ; two coupling groups - more accurate
tau_t = 0.1 0.1 ; time constant, in ps
ref_t = 300 300 ; reference temperature, one for each group, in K
; Pressure coupling is off
pcoupl = no ; no pressure coupling in NVT
; Periodic boundary conditions
pbc = xyz ; 3-D PBC
; Velocity generation
gen_vel = yes ; assign velocities from Maxwell distribution
gen_temp = 300 ; temperature for Maxwell distribution
gen_seed = -1 ; generate a random seed.mdp 文件中的几个主要的参数:
- gen_vel = yes:启动速度生成。使用不同的随机种子 (gen_seed) 会得到不同的初始速度,因此可以从相同的起始结构进行多个(不同的)仿真。
- tcoupl = V-rescale:速度重新定标恒温器是对 Berendsen 弱耦合方法的改进,该方法没有再现正确的动力学系综。 pcoupl = no:不应用压力耦合。
开启NVT
我们将调用 grompp 和 mdrun,就像我们在 EM 步骤中所做的那样:
gmx grompp -f nvt.mdp -c em.gro -r em.gro -p topol.top -o nvt.tpr
gmx mdrun -deffnm nvt分析温度变化
使用下列命令绘图:
gmx energy -f nvt.edr -o temperature.xvg在提示符下键入“16 0”以选择系统的温度并退出。生成的绘图应如下所示: 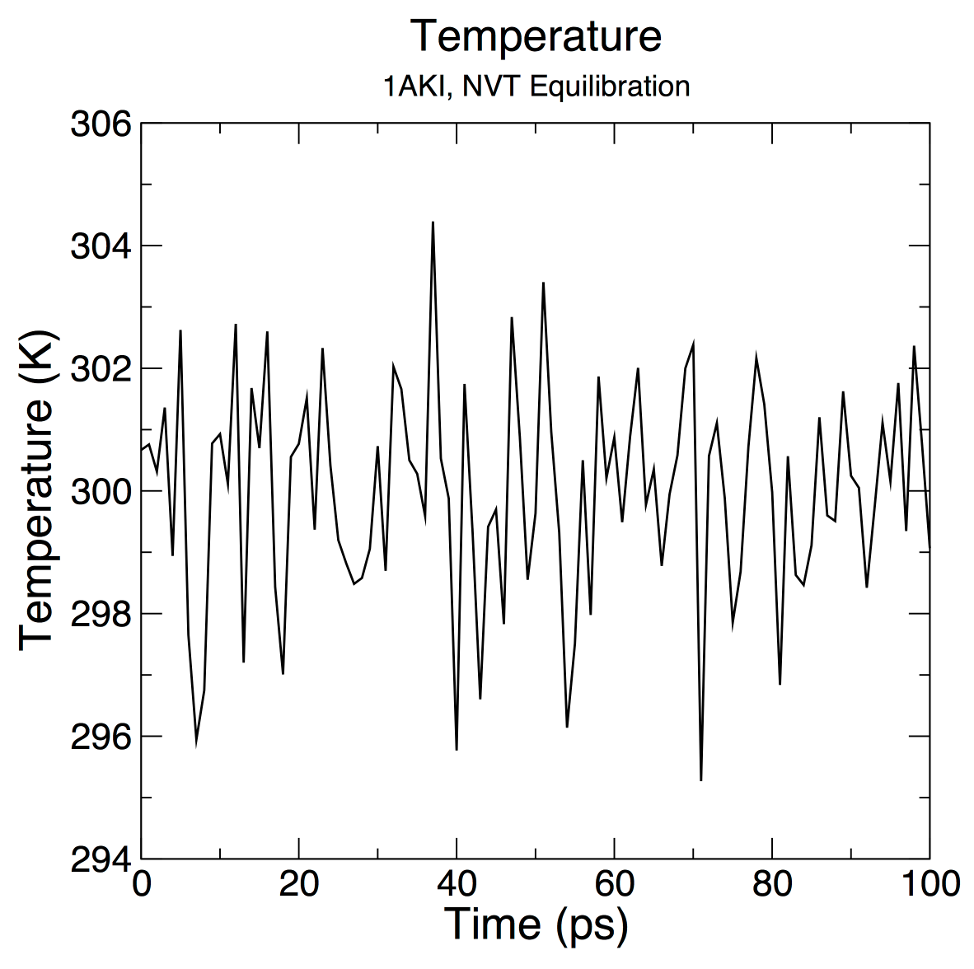
从图中可以清楚地看出,系统的温度很快达到目标值 (300 K),并且在平衡的剩余时间内保持稳定。对于该系统,较短的平衡期(大约 50 ps)可能就足够了。
压力平衡 NPT
上一步 NVT 平衡稳定了系统的温度,下一步开始稳定系统的压力(以及密度)。压力平衡在 NPT 系综下进行,其中粒子数、压力和温度都是恒定的。该系差也称为“等温-等压”系差,与实验条件最相似。
构建mdp文件
构建下列内容的用于 100 ps NPT 平衡的 .mdp 文件:
title = OPLS Lysozyme NPT equilibration
define = -DPOSRES ; position restrain the protein
; Run parameters
integrator = md ; leap-frog integrator
nsteps = 50000 ; 2 * 50000 = 100 ps
dt = 0.002 ; 2 fs
; Output control
nstxout = 500 ; save coordinates every 1.0 ps
nstvout = 500 ; save velocities every 1.0 ps
nstenergy = 500 ; save energies every 1.0 ps
nstlog = 500 ; update log file every 1.0 ps
; Bond parameters
continuation = yes ; Restarting after NVT
constraint_algorithm = lincs ; holonomic constraints
constraints = h-bonds ; bonds involving H are constrained
lincs_iter = 1 ; accuracy of LINCS
lincs_order = 4 ; also related to accuracy
; Nonbonded settings
cutoff-scheme = Verlet ; Buffered neighbor searching
ns_type = grid ; search neighboring grid cells
nstlist = 10 ; 20 fs, largely irrelevant with Verlet scheme
rcoulomb = 1.0 ; short-range electrostatic cutoff (in nm)
rvdw = 1.0 ; short-range van der Waals cutoff (in nm)
DispCorr = EnerPres ; account for cut-off vdW scheme
; Electrostatics
coulombtype = PME ; Particle Mesh Ewald for long-range electrostatics
pme_order = 4 ; cubic interpolation
fourierspacing = 0.16 ; grid spacing for FFT
; Temperature coupling is on
tcoupl = V-rescale ; modified Berendsen thermostat
tc-grps = Protein Non-Protein ; two coupling groups - more accurate
tau_t = 0.1 0.1 ; time constant, in ps
ref_t = 300 300 ; reference temperature, one for each group, in K
; Pressure coupling is on
pcoupl = Parrinello-Rahman ; Pressure coupling on in NPT
pcoupltype = isotropic ; uniform scaling of box vectors
tau_p = 2.0 ; time constant, in ps
ref_p = 1.0 ; reference pressure, in bar
compressibility = 4.5e-5 ; isothermal compressibility of water, bar^-1
refcoord_scaling = com
; Periodic boundary conditions
pbc = xyz ; 3-D PBC
; Velocity generation
gen_vel = no ; Velocity generation is off它与用于 NVT 平衡的参数文件没有太大区别,使用 Parrinello-Rahman 恒压器添加了压力耦合部分,其他一些更改如下:
- continuation = yes:我们从 NVT 平衡阶段继续模拟
- gen_vel = no:从轨迹中读取速度(见下文)
开启NPT
随后继续调用 grompp 和 mdrun,和NVT相同。请注意,我们现在包括 -t 标志以包含 NVT 平衡的检查点文件;此文件包含继续模拟所需的所有状态变量。为了守恒 NVT 期间产生的速度,我们必须包含此文件。坐标文件 (-c) 是 NVT 仿真的最终输出。
gmx grompp -f npt.mdp -c nvt.gro -r nvt.gro -t nvt.cpt -p topol.top -o npt.tpr
gmx mdrun -deffnm npt分析结果
先使用energy来分析压力级数:
gmx energy -f npt.edr -o pressure.xvg在提示符处键入 “18 0” 以选择系统的压力并退出。生成的绘图应如下所示: 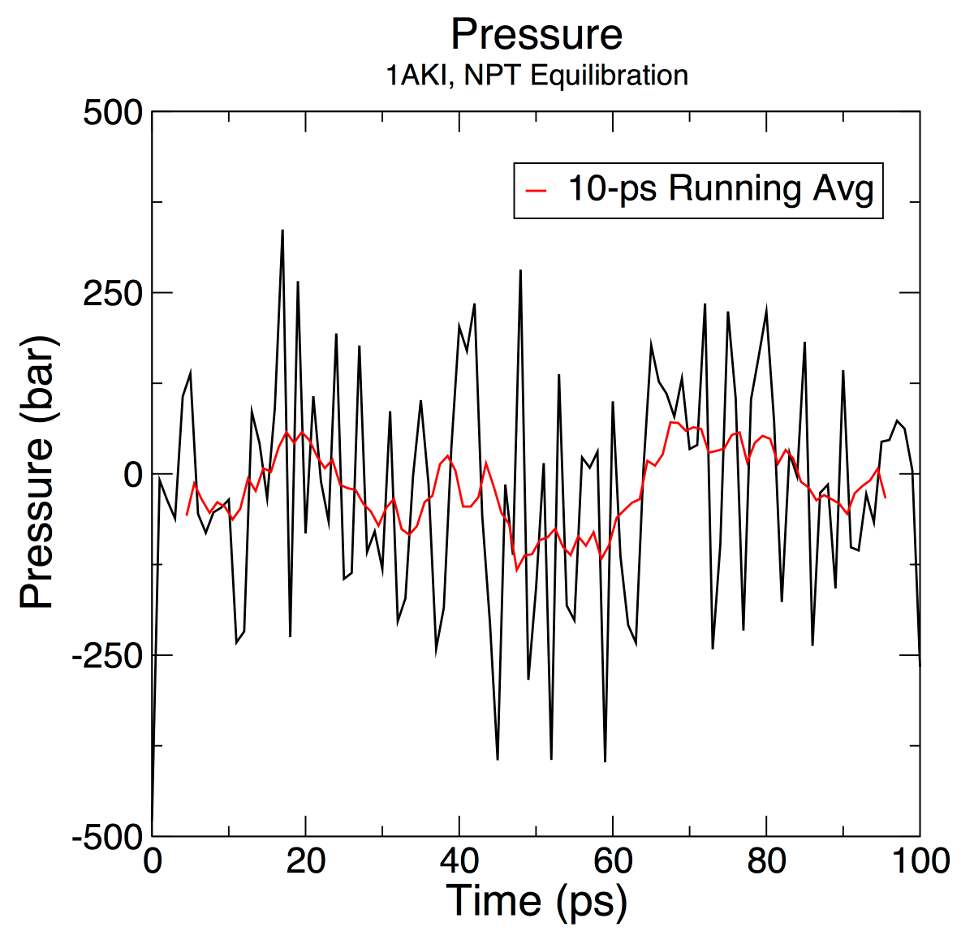
压力值在 100 ps 平衡阶段波动很大,但这种行为并不意外。这些数据的运行平均值在图中绘制为红线。在平衡过程中,压力的平均值为 7.5 ± 160.5 bar。请注意,参考压力设置为 1 bar,那么这个结果是否可以接受?压力是在 MD 模拟过程中波动很大的量,从较大的均方根波动 (160.5 bar) 中可以清楚地看出,因此从统计学上讲,无法区分获得的平均值(7.5 ± 160.5 bar)和目标/参考值(1 bar)之间的差异。
再分析密度,这次使用energy并在提示符处输入 “24 0”。
gmx energy -f npt.edr -o density.xvg与压力一样,密度的运行平均值也用红色绘制。100 ps 过程中的平均值为 1019 ± 3 kg m-3,接近 1000 kg m-3 的实验值和 SPC/E 模型的预期密度 1008 kg m-3。SPC/E 水模型的参数与水的实验值非常相似。密度值随着时间的推移非常稳定,表明系统现在在压力和密度方面得到了很好的平衡。 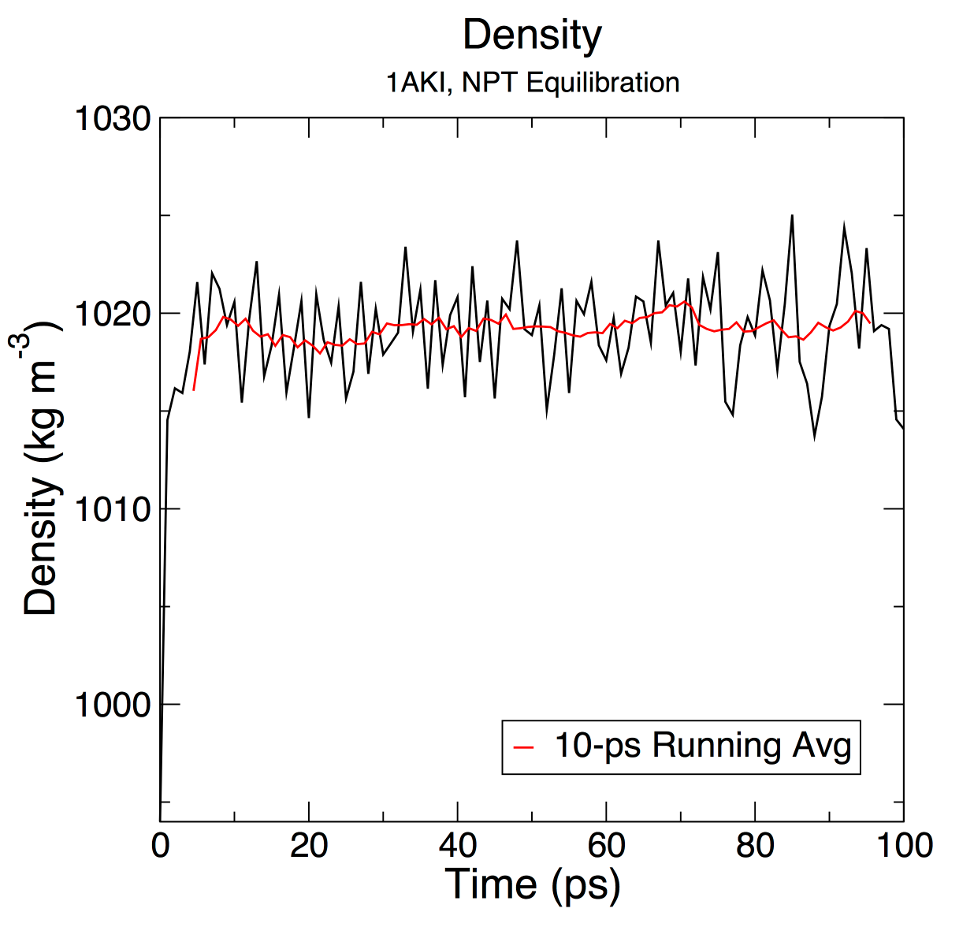
如果获得的密度值与该结果不匹配,可能是因为与压力相关的项收敛速度较慢,因此可能需要运行 NPT 平衡的时间略长于此处指定的时间。
开始模拟
完成两个平衡阶段后,系统现在在所需的温度和压力下得到很好的平衡。现在,我们已准备好释放位置约束并运行生产 MD 进行数据收集。这个过程就像我们之前看到的一样,因为我们将使用 checkpoint 文件(在本例中现在包含 preserve 压力耦合信息)到 grompp。我们将运行 1 ns 的 MD 仿真。
mdp文件
构建下列的md.mdp配置文件:
title = OPLS Lysozyme NPT equilibration
; Run parameters
integrator = md ; leap-frog integrator
nsteps = 500000 ; 2 * 500000 = 1000 ps (1 ns)
dt = 0.002 ; 2 fs
; Output control
nstxout = 0 ; suppress bulky .trr file by specifying
nstvout = 0 ; 0 for output frequency of nstxout,
nstfout = 0 ; nstvout, and nstfout
nstenergy = 5000 ; save energies every 10.0 ps
nstlog = 5000 ; update log file every 10.0 ps
nstxout-compressed = 5000 ; save compressed coordinates every 10.0 ps
compressed-x-grps = System ; save the whole system
; Bond parameters
continuation = yes ; Restarting after NPT
constraint_algorithm = lincs ; holonomic constraints
constraints = h-bonds ; bonds involving H are constrained
lincs_iter = 1 ; accuracy of LINCS
lincs_order = 4 ; also related to accuracy
; Neighborsearching
cutoff-scheme = Verlet ; Buffered neighbor searching
ns_type = grid ; search neighboring grid cells
nstlist = 10 ; 20 fs, largely irrelevant with Verlet scheme
rcoulomb = 1.0 ; short-range electrostatic cutoff (in nm)
rvdw = 1.0 ; short-range van der Waals cutoff (in nm)
; Electrostatics
coulombtype = PME ; Particle Mesh Ewald for long-range electrostatics
pme_order = 4 ; cubic interpolation
fourierspacing = 0.16 ; grid spacing for FFT
; Temperature coupling is on
tcoupl = V-rescale ; modified Berendsen thermostat
tc-grps = Protein Non-Protein ; two coupling groups - more accurate
tau_t = 0.1 0.1 ; time constant, in ps
ref_t = 300 300 ; reference temperature, one for each group, in K
; Pressure coupling is on
pcoupl = Parrinello-Rahman ; Pressure coupling on in NPT
pcoupltype = isotropic ; uniform scaling of box vectors
tau_p = 2.0 ; time constant, in ps
ref_p = 1.0 ; reference pressure, in bar
compressibility = 4.5e-5 ; isothermal compressibility of water, bar^-1
; Periodic boundary conditions
pbc = xyz ; 3-D PBC
; Dispersion correction
DispCorr = EnerPres ; account for cut-off vdW scheme
; Velocity generation
gen_vel = no ; Velocity generation is off执行MD模拟
下列命令可以将模拟参数、初始结构、拓扑信息和检查点文件整合成一个二进制文件(.tpr 文件),供后续的分子动力学模拟使用:
gmx grompp -f md.mdp -c npt.gro -t npt.cpt -p topol.top -o md_0_1.tprgrompp 将打印 PME 负载的估计值,这将决定应该有多少个处理器专用于 PME 计算,以及有多少个处理器用于 PP 计算。
对于立方体盒子,最佳设置的PME负载为0.25(3:1 PP:PME);对于十二面体箱,最佳PME载荷为0.33(2:1 PP:PME)。
执行mdrun时,程序应自动确定为PP和PME计算分配的最佳处理器数量。因此,请确保为计算指定适当数量的线程/内核(-nt X的值),以便获得最佳性能。
执行 mdrun:
gmx mdrun -deffnm md_0_1 -v使用-v命令会显示每一步的具体进度
gmx mdrun -deffnm md_0_1 -v -cpi md_0_1.cpt 12000.cpt 文件包含前一阶段模拟的状态信息(如速度、能量等),用于从断点继续模拟,使用-cpi命令,可以从第12000步重新接续计算。
GPU加速
在 GROMACS 2018 中,可以将 PME 计算卸载到图形处理单元 (GPU) 中,从而大大加快仿真速度。使用 Titan Xp GPU,该系统可以以惊人的 295 ns/天的速度进行模拟!
假设您有一个可用的 GPU,使用它的 mdrun 命令非常简单:
gmx mdrun -deffnm md_0_1 -nb gpu结果分析
现在我们已经模拟了蛋白质,我们应该对系统进行一些分析。在本教程中,将介绍一些基本动力学模拟的数据分析工具。
trjconv校正
trjconv 用作后处理工具,用于去除坐标、校正周期性或手动更改轨迹(时间单位、帧频率等)。我们使用 trjconv 来考虑系统中的任何周期性,蛋白质将通过晶胞扩散,并且可能看起来“破碎”或可能“跳”到盒子的另一侧。要考虑此类行为,请发出以下内容:
gmx trjconv -s md_0_1.tpr -f md_0_1.xtc -o md_0_1_noPBC.xtc -pbc mol -center选择 1 (“Protein”) 作为要居中的组,选择 0 (“System”) 作为输出。我们将根据这个 “修正” 的轨迹进行所有分析。让我们先看看结构稳定性。
RMSD
GROMACS 有一个用于 RMSD 计算的内置实用程序,称为 rms,使用以下命令:
gmx rms -s md_0_1.tpr -f md_0_1_noPBC.xtc -o rmsd.xvg -tu ns选择 4(“Backbone”)作为最小二乘拟合和 RMSD 计算的组。-tu 标志将以 ns 为单位输出结果,即使轨迹以 ps 为单位编写。这样做是为了使输出清晰(特别是如果你有一个长时间的模拟 - 1e+05 ps 看起来不如 100 ns)。
使用下列命令来计算相对于晶体结构的 RMSD:
gmx rms -s em.tpr -f md_0_1_noPBC.xtc -o rmsd_xtal.xvg -tu ns将两个结果一起绘制,结果如下所示: 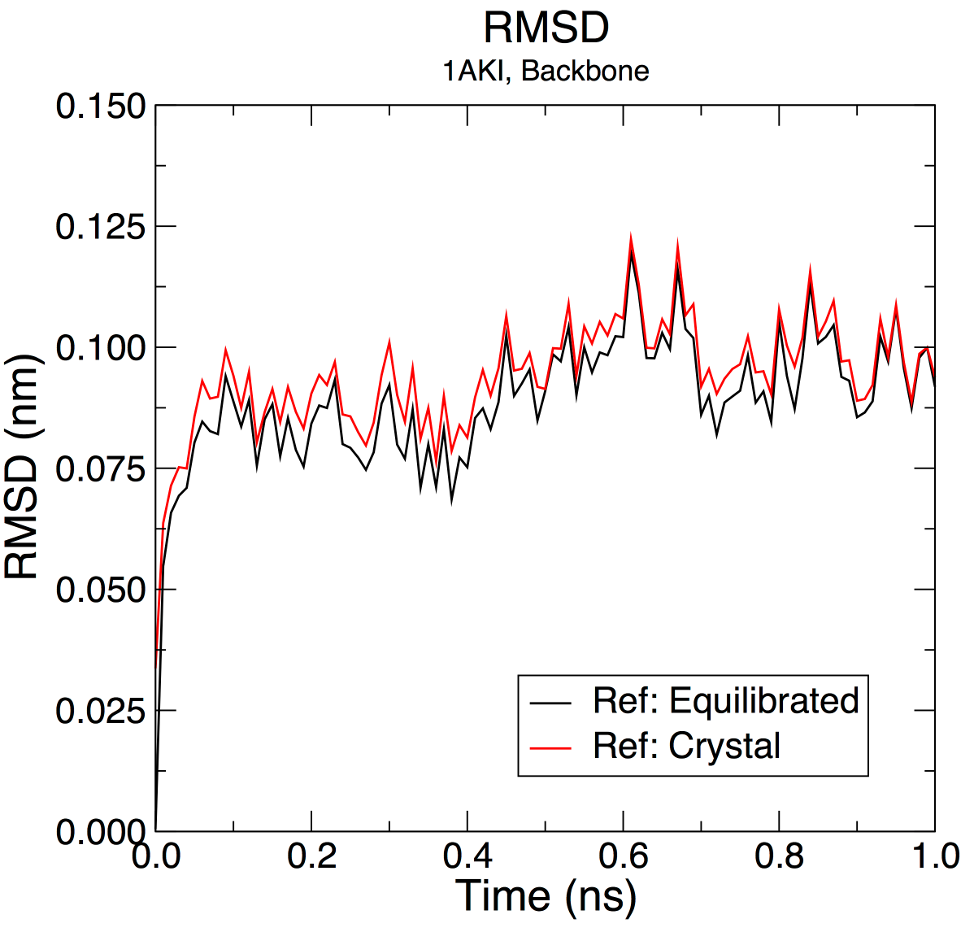
两个时间序列都显示 RMSD 水平低至 ~0.1 nm (1 Å),表明结构非常稳定。图之间的细微差异表明,t = 0 ns 处的结构与这种晶体结构略有不同。这是意料之中的,因为它已经被能量最小化,而且如前所述,位置约束并不是 100% 完美的。
回转半径
蛋白质的回转半径是其紧凑性的量度。如果蛋白质稳定折叠,它可能会保持相对稳定的 Rg 值。如果蛋白质去折叠,它的 Rg 会随着时间的推移而变化。让我们在模拟中分析溶菌酶的回转半径:
gmx gyrate -s md_0_1.tpr -f md_0_1_noPBC.xtc -o gyrate.xvg选择第 1 组 (Protein) 进行分析: 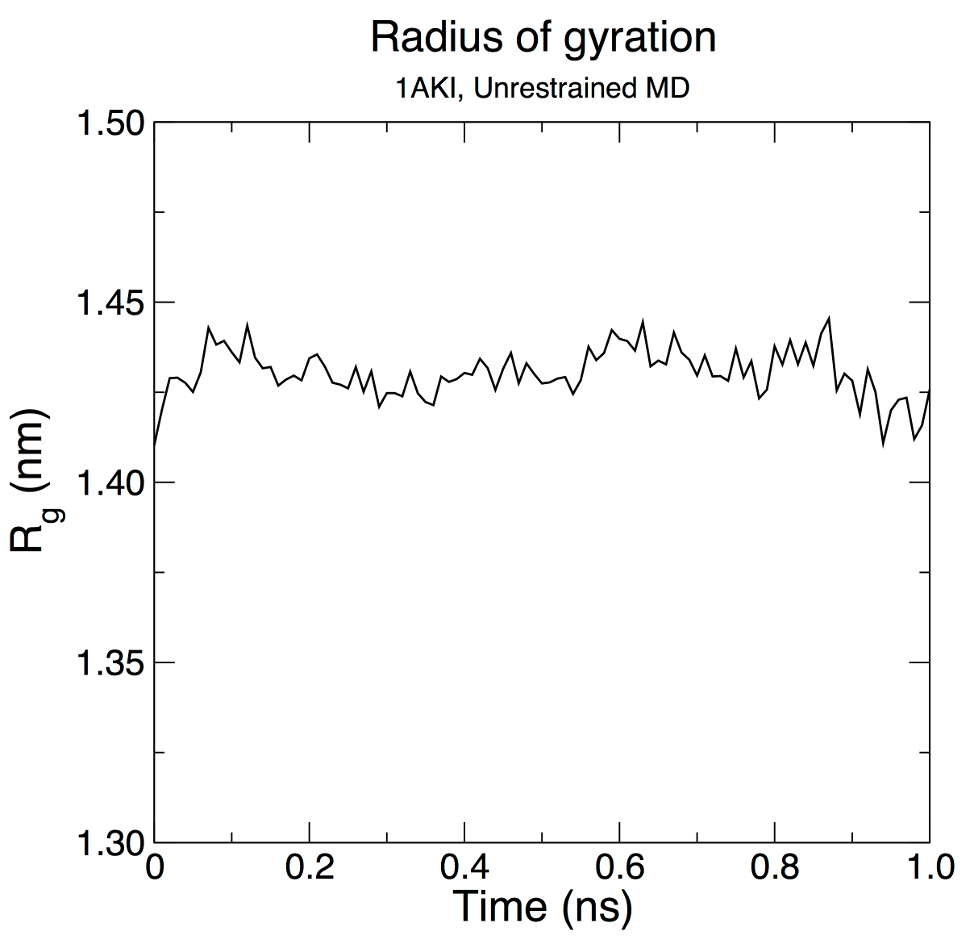
从合理不变的 Rg 值中可以看出,蛋白质在 300 K 下以紧凑(折叠)形式在 1 ns 的过程中保持非常稳定。
总结
现在,您已经使用 GROMACS 进行了分子动力学模拟,并分析了一些结果。不应将本教程视为全面。使用 GROMACS 可以进行更多类型的模拟(自由能计算、非平衡 MD 和正则模态分析,仅举几例)。您还应该查看文献和 GROMACS 手册,以调整此处提供的.mdp文件,以提高效率和准确性。